마이크로바이옴(microbiome) 연구는 크게 연구설계, 실험, 통계분석 세 단계로 진행됩니다. 각 단계를 어떻게 진행할 지에 대한 결정은 최종적인 연구결과에 상당한 영향을 미칩니다. 이번 내용에서는 성공적인 마이크로바이옴 연구를 위해 이 세가지 단계 중 가장 중요한 연구설계 및 실험 단계에서 고려해야 할 몇 가지 부분들을 살펴보고자 합니다.
1. 연구 설계
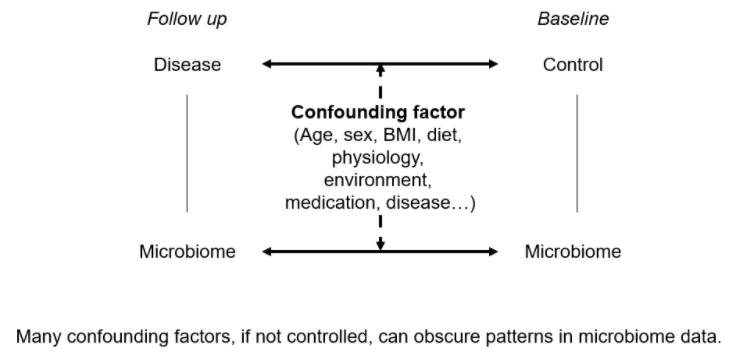
마이크로바이옴 연구는 크게 환자-대조군(case-control) 연구와 종적개입연구(longitudinal intervention study)로 나눌 수 있습니다. 두 가지 연구 모두 각각 대조군 및 기준선과 비교하여 환자 및 추적검사 검체에서 microbiome community structure/composition에 어떠한 차이가 있는지를 밝히는데 초점이 있습니다. 여기서 가장 중요한 부분은 연구 대상자를 모집할 때 교란요인(confounding factor)을 잘 통제해주어야 한다는 것입니다. 교란용인이란 독립변수(dependent variable)와 비독립년수(independent variable) 모두에 영향을 끼치는 인자로써 적절히 통제되지 않으면 독립변수와 비독립변수 간에 거짓된 연관성을 나타낼 수 있습니다.
마이크로바이옴 연구에서도 아래 그림과 같이 나이, 성별, BMI뿐만 아니라 식이, 약물, 동반 질환력 등 다양한 요소들이 질환의 유무(dependent variable)뿐 아니라 microbiome(independent variable)에도 동시에 영향을 미치므로 환자와 대조군 간에 이러한 confounding factor를 잘 통제해주어야 실제로 질환과 연관되는 정확한 마이크로바이옴 표지자를 찾을 수 있습니다. 하지만 모든 교란요인을 통제하기는 현실적으로 불가능하며 일반적으로 나이, 성별, BMI, 약물복용 유무, 질환 유무 정도를 통제하고 그 외의 요소들은 분석시 통계적 기법을 이용하여 보정해 주기도 합니다.
한 가지 더 고려할 부분은 검체 수 선정입니다. 일반적인 연구시 검체 수는 환자-대조군 간 차이의 정도(effect size)와 통계적 검증력(statistical power)를 고려하여 선정합니다. 하지만 아직까지 마이크로바이옴 분야에서는 이러한 effect size나 statistical power에 대한 연구가 미흡한 실정입니다. 따라서 마이크로바이옴 연구에서는 기존의 연구를 참고하여 검체 수를 산정하는 방식이 가장 권고되나 마이크로바이옴의 특성상 개인간생물학적 변이가 상당히 크므로 가능한 많은 수의 검체를 모집하는 것이 좋겠습니다.
2. 실험
연구설계, 실험, 통계분석 중 마이크로바이옴 연구결과에 가장 큰 영향을 끼치는 부분은 실험 단계입니다. 마이크로바이옴 실험방법은 저렴한 비용 때문에 주로 16S rRNA amplicon sequencing 방법으로 진행되며, 결과는 검체 내 존재하는 마이크로바이옴의 구성과 각각의 비율(composition and proportion)로 표현됩니다. 16S amplicon sequencing을 이용한 마이크로바이옴 실험결과는 아래와 같이 검체 채취 및 운송, DNA 추출, PCR, 시퀀싱, BI 분석까지 매우 다양한 과정에 영향을 받으며, 따라서 여러 과정 중 일부 방법이 바뀌어도 실험결과 즉, 검출되는 미생물의 비율이나 검출 유무가 크게 달라질 수 있습니다.
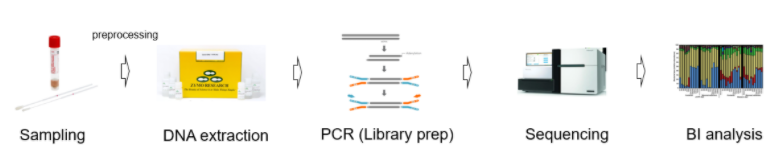
아직까지 마이크로바이옴 실험과정에 대한 표준화된 지침은 없고 현재의 마이크로바이옴 연구에는 매우 다양한 실험 프로토콜이 적용되고 있습니다. 이에 연구마다 상이한 결과가 많이 도출되고 있는 실정이나, 성공적인 마이크로바이옴 연구를 위해서는 가장 최적의 실험 프로토콜을 선정하는 것이 중요합니다. 여기서 최적의 실험 프로토콜이라고 함은 실제 샘플에 존재하는 마이크로바이옴 구성과 비율을 가장 정확하게 반영할 수 있는 방법을 의미합니다.
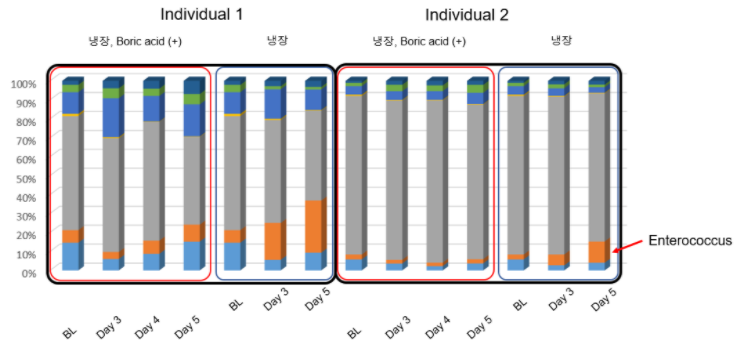
몇 가지 예시를 보여드리겠습니다. 아래는 boric acid를 첨가하여 냉장 보관한 소변 검체와 boric acid 없이 냉장 보관한 소변 검체의 시간에 따른 마이크로바이옴 결과 추이를 나타낸 그래프입니다. 보시는 바와 같이 boric acid를 첨가한 소변에 비해 boric acid를 첨가하지 않은 소변에서는 시간에 따라 enterococcus가 증식하는 경향이 뚜렷이 나타나는 것을 확인할 수 있습니다.
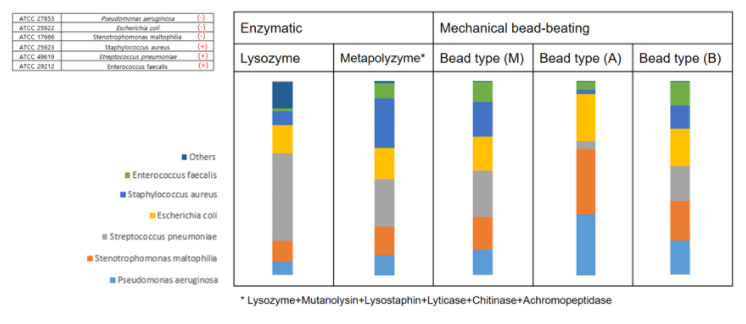
아래는 6개의 그람 양성 박테리아와 그람 음성 박테리아로 구성된 표준균주를 이용하여 DNA 추출 방법에 따른 결과를 비교한 것입니다. 미생물의 DNA 추출시 핵심은 그람 양성 박테리아의 두꺼운 펩티도글리칸(peptidoglycan) 세포벽을 잘 파괴하는 것입니다. 펩티도글리칸 세포벽을 파괴하는 방법에는 효소를 이용하는 화학적인 방법과 비드비팅(bead-beating)을 이용하는 물리적인 방법이 있는데, 효소나 비드 형태, 비드비팅 시간을 어떻게 선택하느냐에 따라 결과가 매우 상이하게 나타날 수 있습니다. DNA 추출 단계에서 추출이 제대로 이루어지지 않으면 그 뒤의 실험이나 분석을 아무리 잘 수행하더라도 잘못된 결과가 도출될 수 밖에 없을 것입니다.
또한 아래와 같이 16S variable region 중 어떠한 부분을 증폭하는지에 따라서도 결과가 달라질 수 있는데, 이는 모든 박테리아에 결합할 수 있는 최선의 보편적 시발체(universal primer)를 디자인 할지라도 박테리아마다 해당 부위 염기서열이 조금씩 다르기 때문에 시발체가 수많은 종류의 박테리아를 동일한 효율로 결합하여 증폭시키는 것은 불가능하기 때문입니다. 따라서, 다양한 박테리아로 구성된 마이크로바이옴 검체에 서로 다른 종류의 시발체를 이용하여 PCR을 시행하면 시발체가 가진 고유의 박테리아 증폭 효율에 따라 구성과 비율이 조금씩 달라질 수 있으며, 심지어는 특정 시발체에서는 특정 박테리아를 검출하지 못할 수도 있습니다.
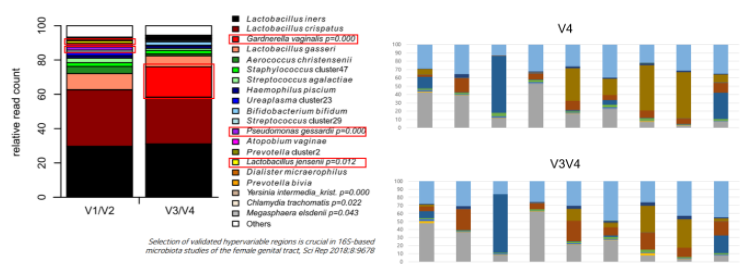
지난 십여년간 마이크로바이옴 연구가 비약적으로 확대되었지만 마이크로바이옴이 임상 영역으로 진입하기 위해서는 아직도 해결해야 할 많은 과제가 있으며, 그 중 하나가 실험방법의 표준화에 대한 부분입니다. 그 전까지는 각 실험방법의 특성과 장단점을 정확하게 인지하고 해석하는 지혜가 요구됩니다.
녹십자지놈 송주선


